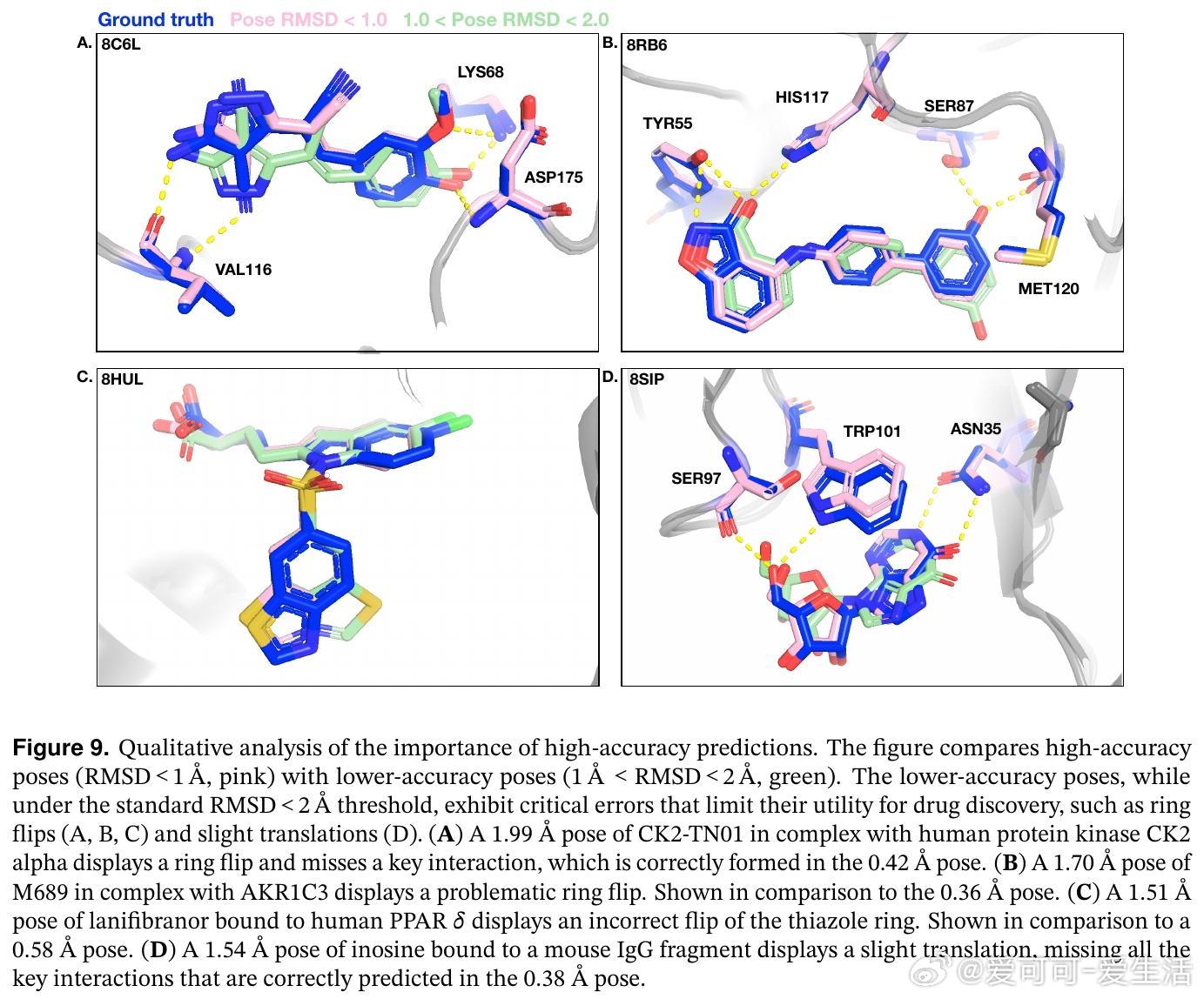
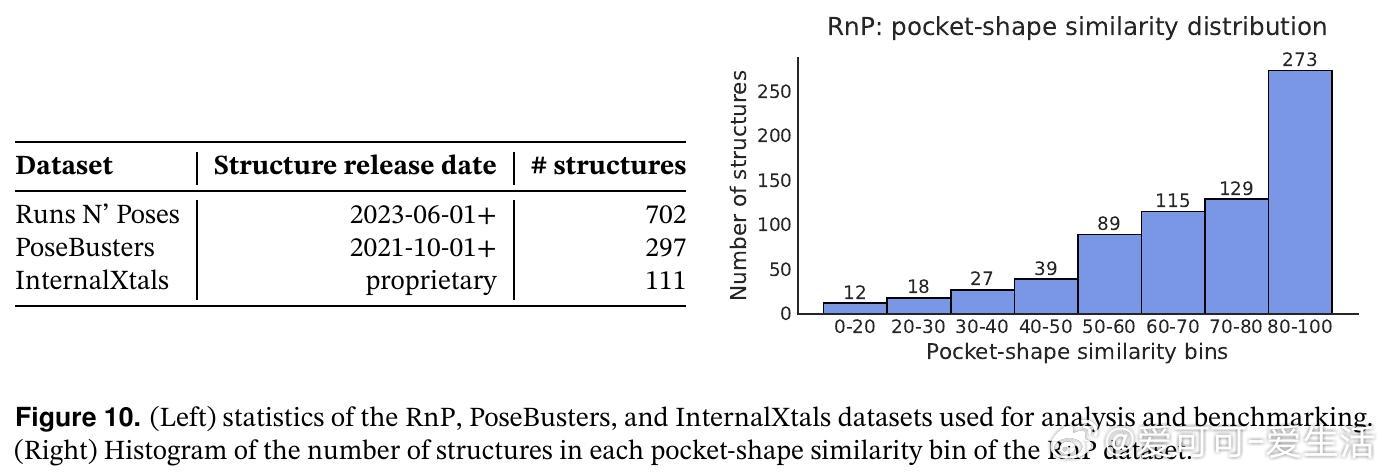
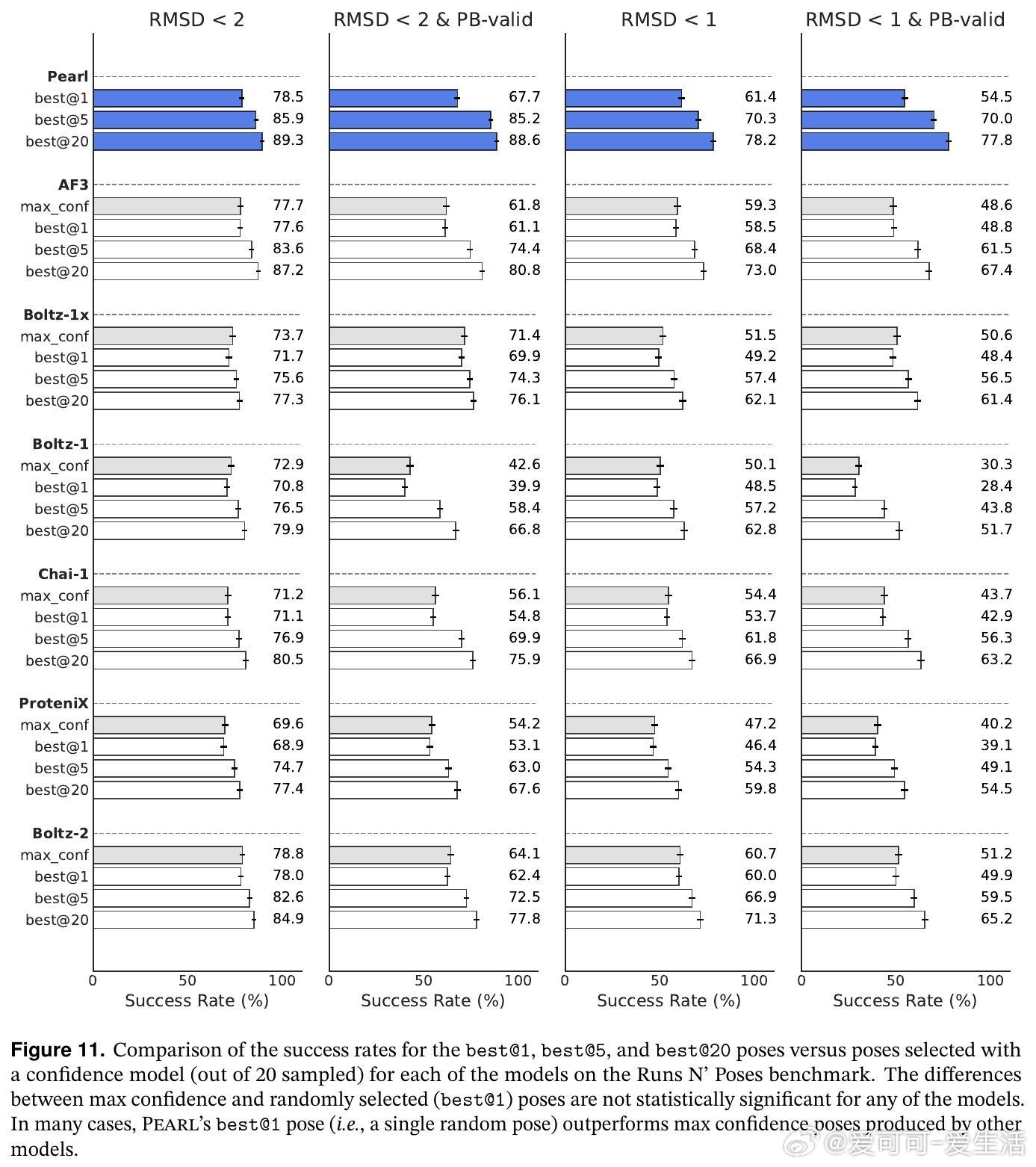
[LG]《Pearl: A Foundation Model for Placing Every Atom in the Right Location》G R T A Dobles, N Jovic, K Leidal, P Murugan... [Genesis Molecular AI] (2025)
Pearl:每个原子精准定位的基础模型,推动蛋白-配体结构预测新高度
蛋白-配体复合物三维结构的准确预测,是计算机辅助药物设计中的核心难题。传统物理对接方法受限于蛋白刚性假设,难以捕捉诱导适应效应;而早期蛋白折叠模型则无法联合预测配体结构。近期深度学习模型如AlphaFold 3实现了多分子共折叠,开创了新纪元,但仍面临数据稀缺、物理合理性不足和推断时灵活性有限等挑战。
针对这些问题,Genesis Molecular AI团队提出了Pearl(Placing Every Atom in the Right Location)模型,具有以下三大创新:
1. 大规模合成数据训练策略,显著缓解蛋白质结构数据库(PDB)中数据稀缺和偏倚,提升模型泛化能力;
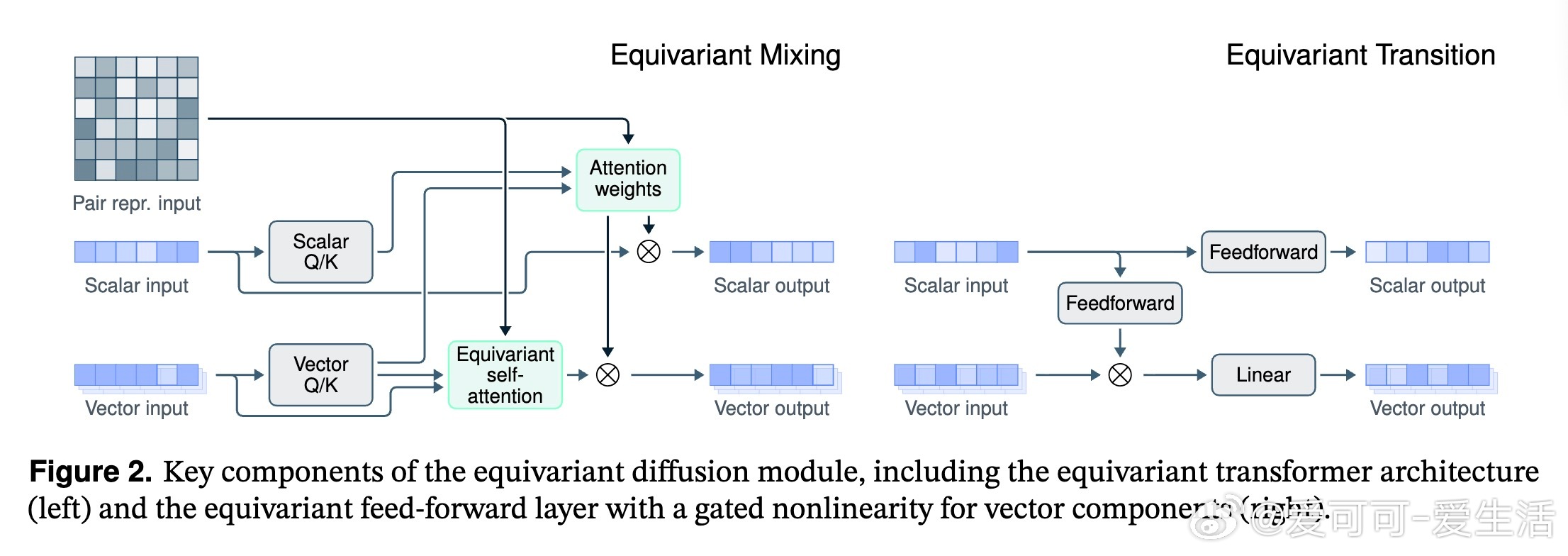
2. 引入SO(3)-等变扩散模块,内置三维空间旋转对称性,提升训练效率和预测的物理合理性;
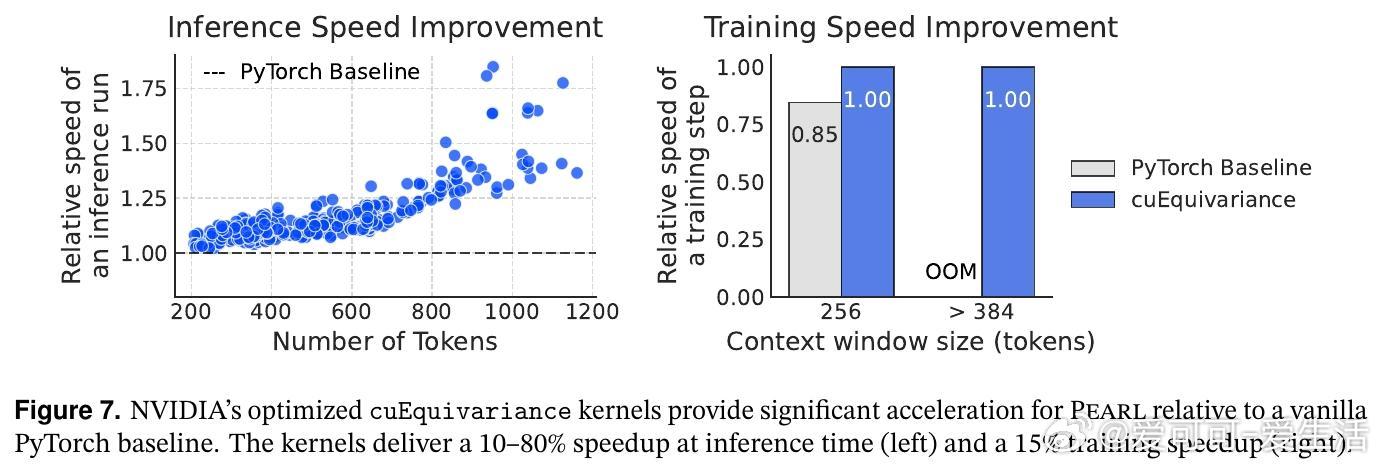
3. 多链模板系统支持蛋白与非聚合物组分联合建模,实现条件与无条件两种推断模式,极大增强推断时的可控性和灵活性。
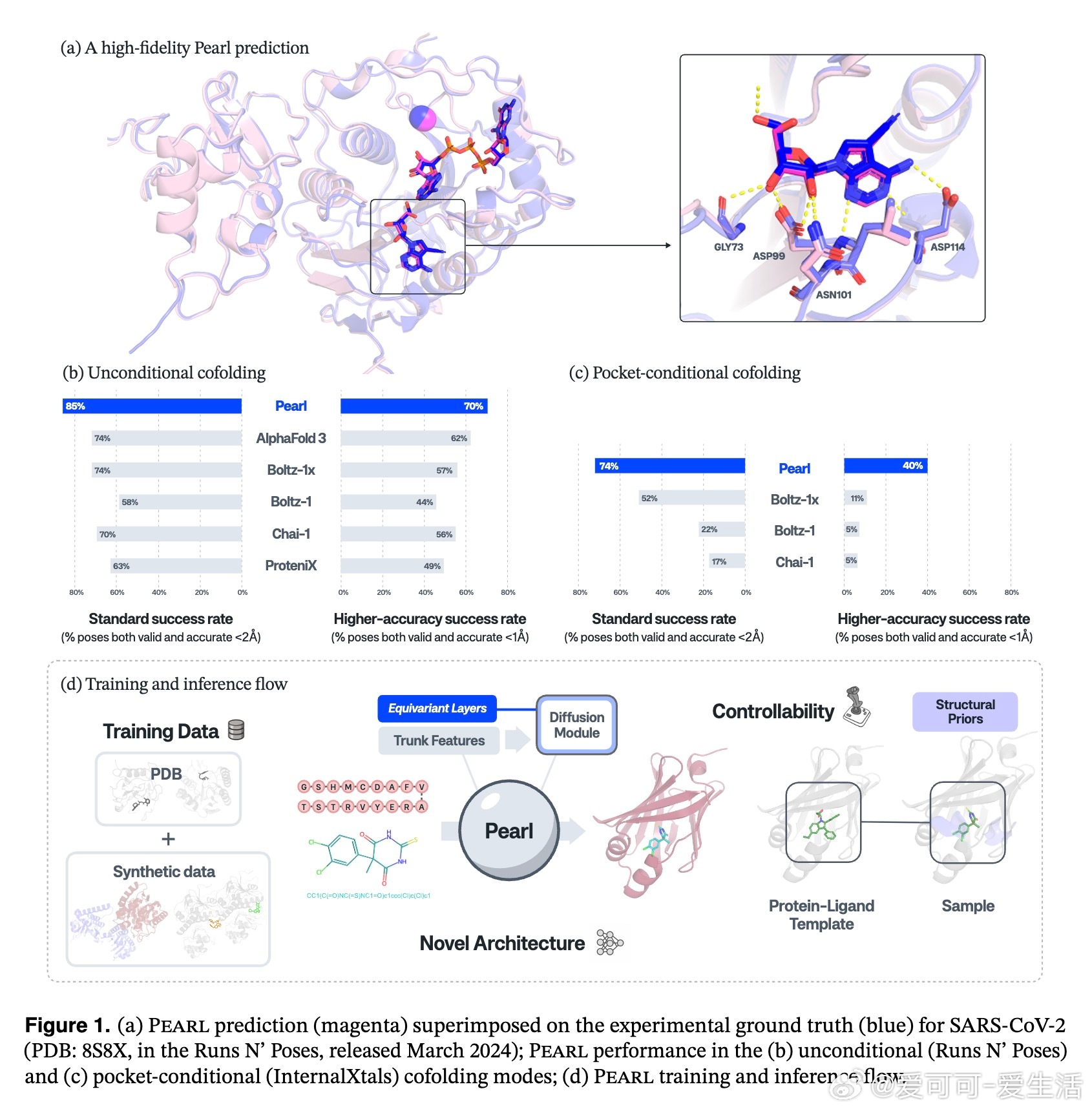
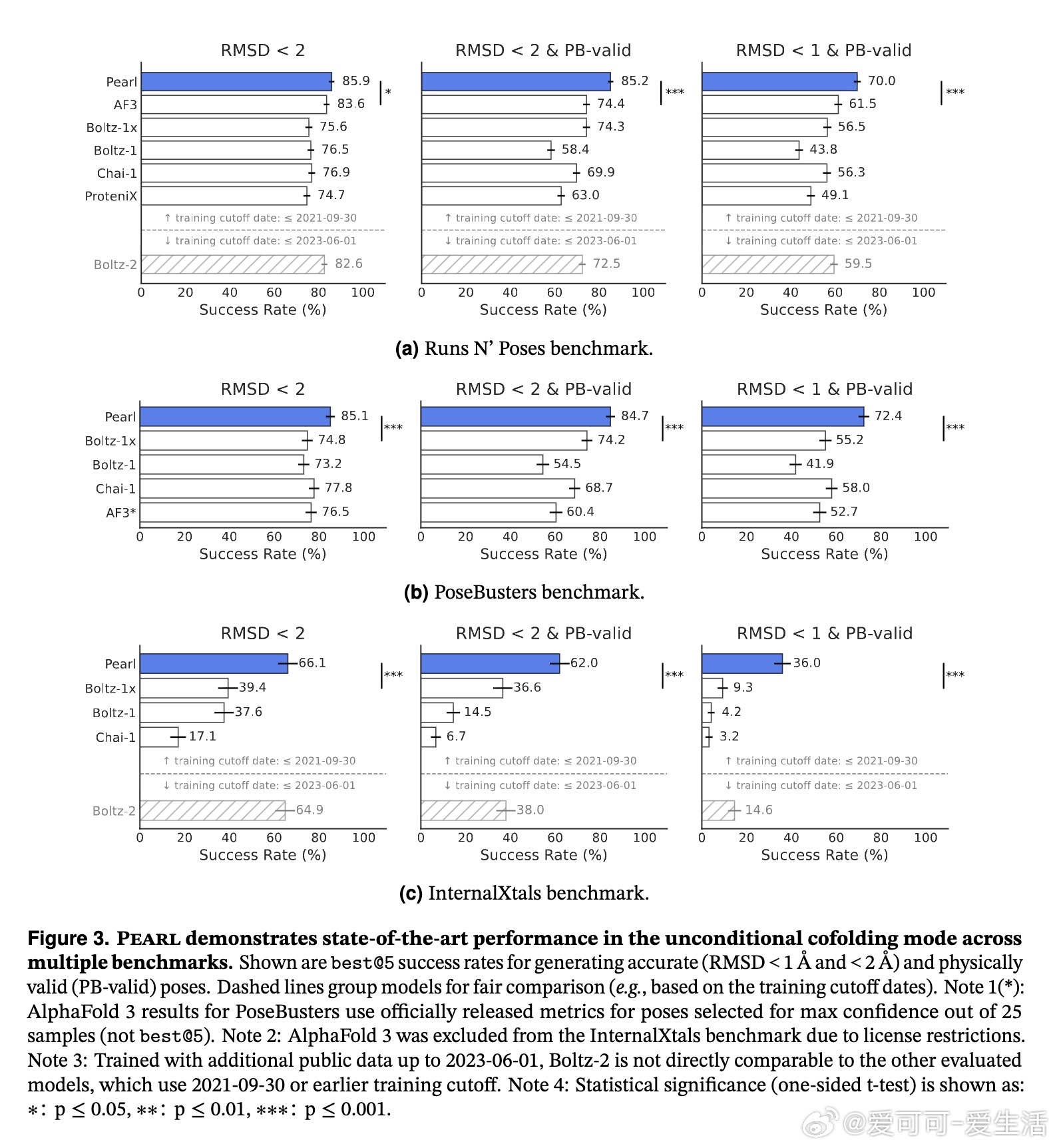
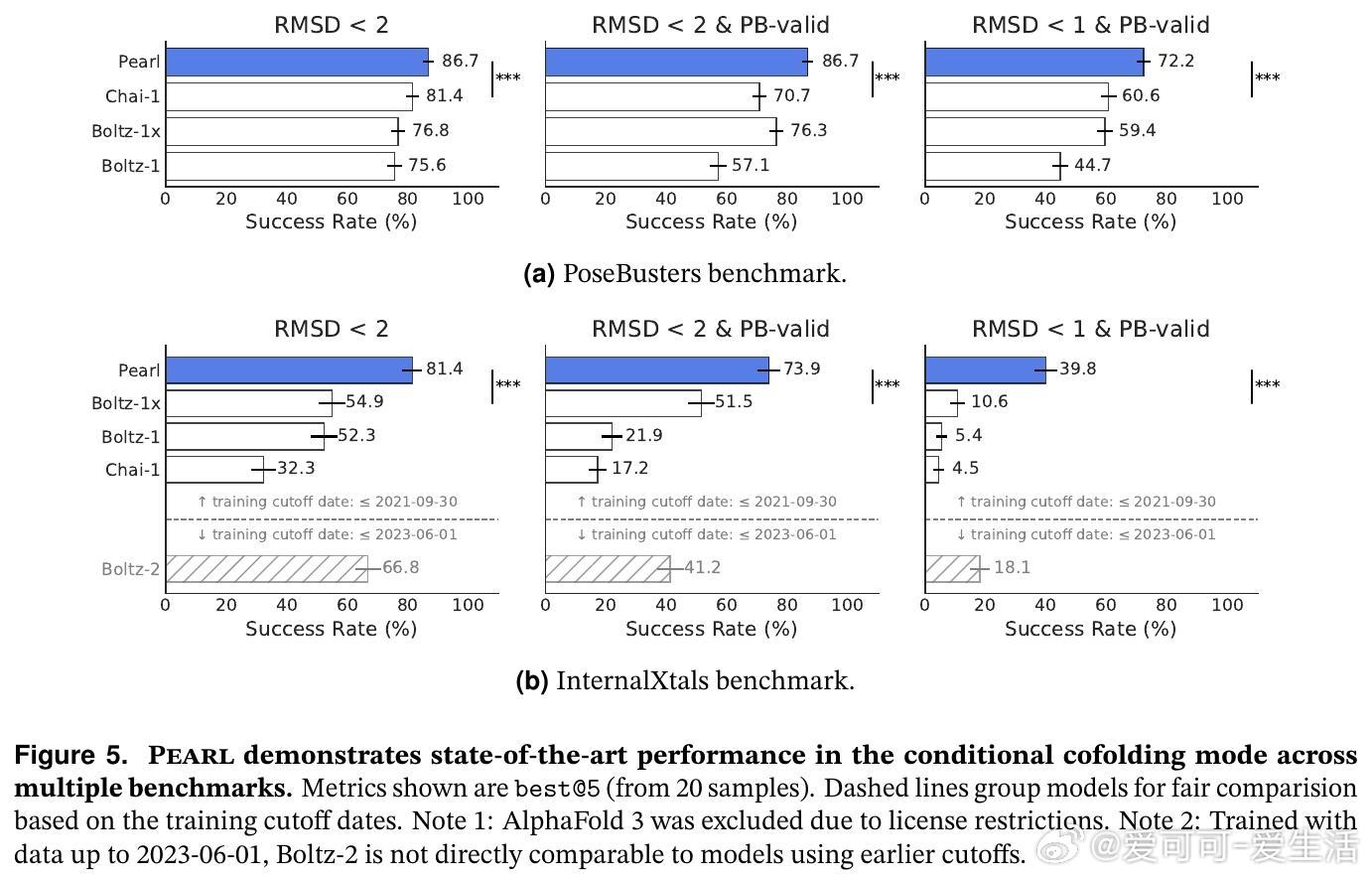
Pearl在多个公开基准(Runs N’ Poses、PoseBusters)以及专有真实药物靶点集(InternalXtals)中均实现新一代领先表现:
- 在生成精准(RMSD < 2Å)且物理有效的构象上,较AlphaFold 3提升约14%;
- 在更严格的高精度阈值(RMSD < 1Å)下,对于真实药物靶点,Pearl表现几乎是现有最优模型的4倍;
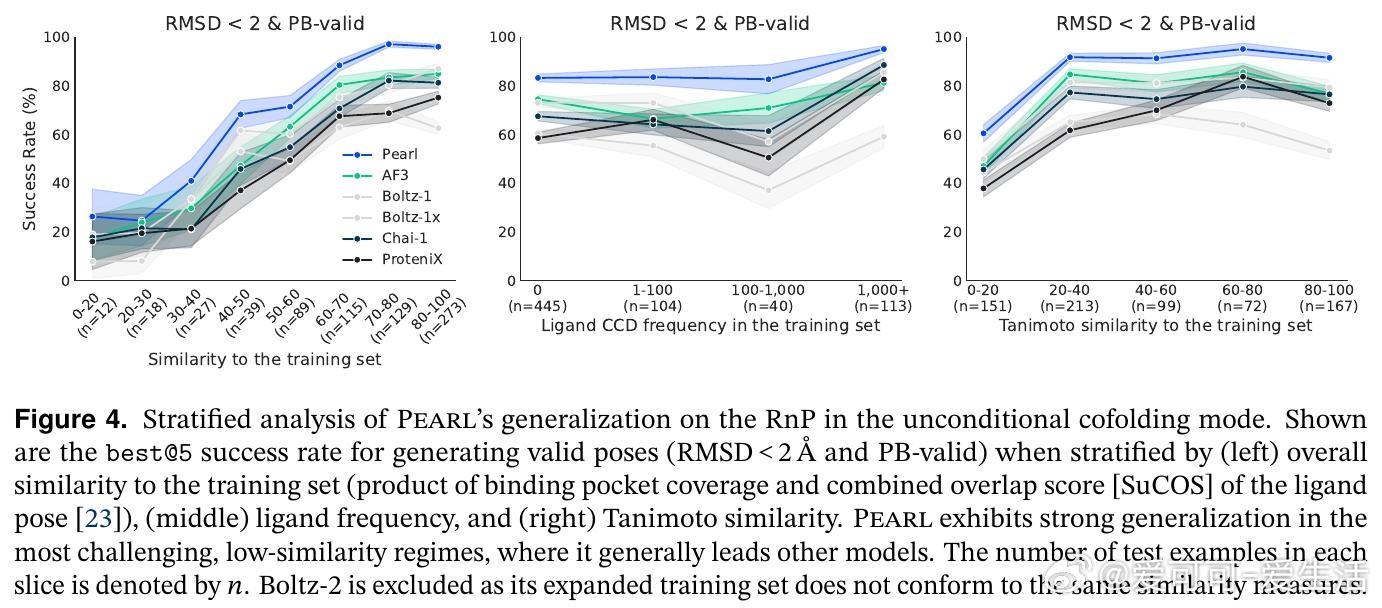
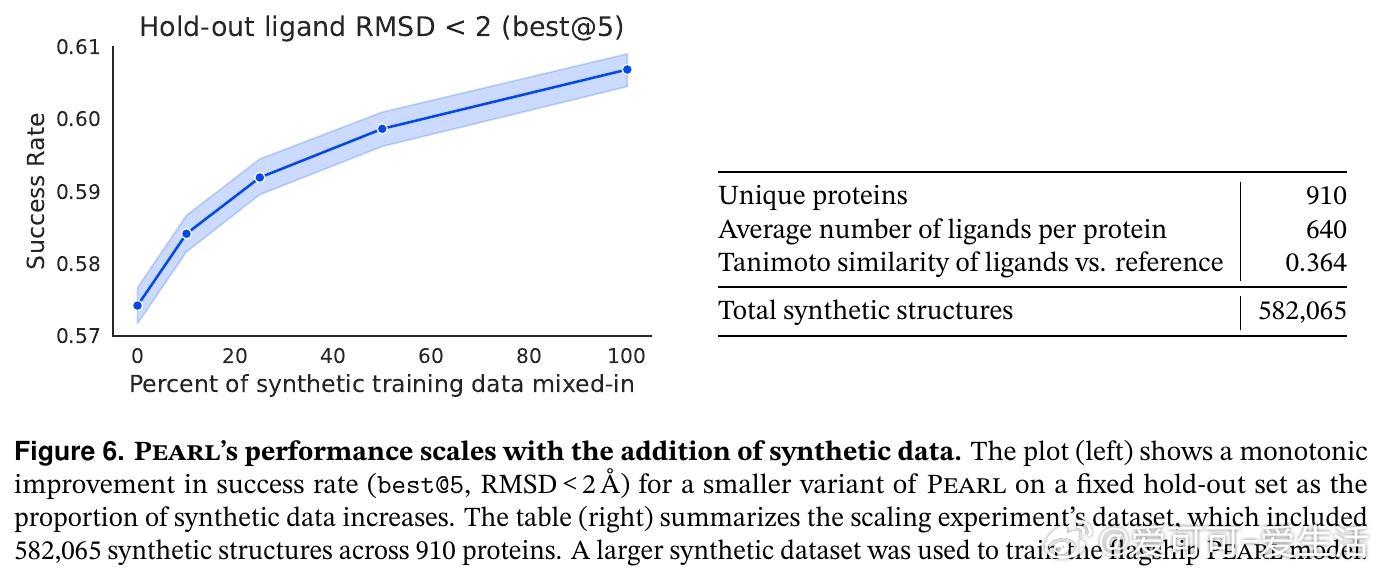
- 合成数据规模与模型性能呈单调正相关,验证了合成数据扩展训练集的重要性。
此外,Pearl的高准确率显著提升了药物化学设计的实用价值,能够为结构导向的药物发现提供更可靠的构象预测和指导。模型的多链模板设计允许科研人员灵活利用已知结构信息,进一步推动药物设计效率。
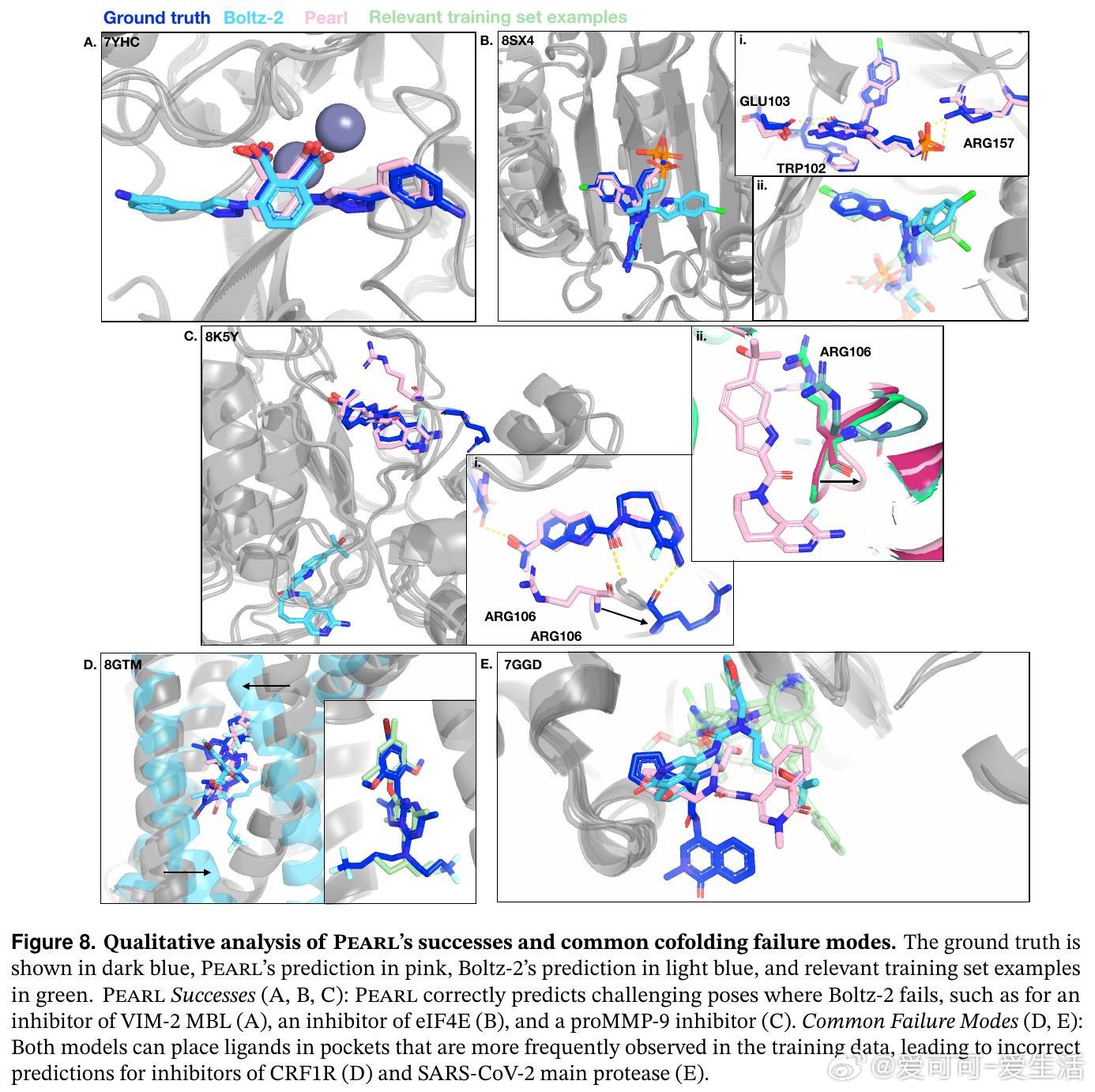
尽管Pearl在泛化、物理合理性和高准确率方面取得突破,依然存在针对极端构象和罕见诱导适应的挑战,未来工作将聚焦于提升远离训练分布样本的鲁棒性和开发更精确的构象筛选机制。
总结来看,Pearl通过创新的数据增强、结构等变设计和灵活的模板条件机制,定义了蛋白-配体共折叠的新范式,为AI驱动的药物发现和科学生成模型开辟了广阔前景。
全文阅读及数据详解请见:
arxiv.org/abs/2510.24670